文 | 追问nextquestion
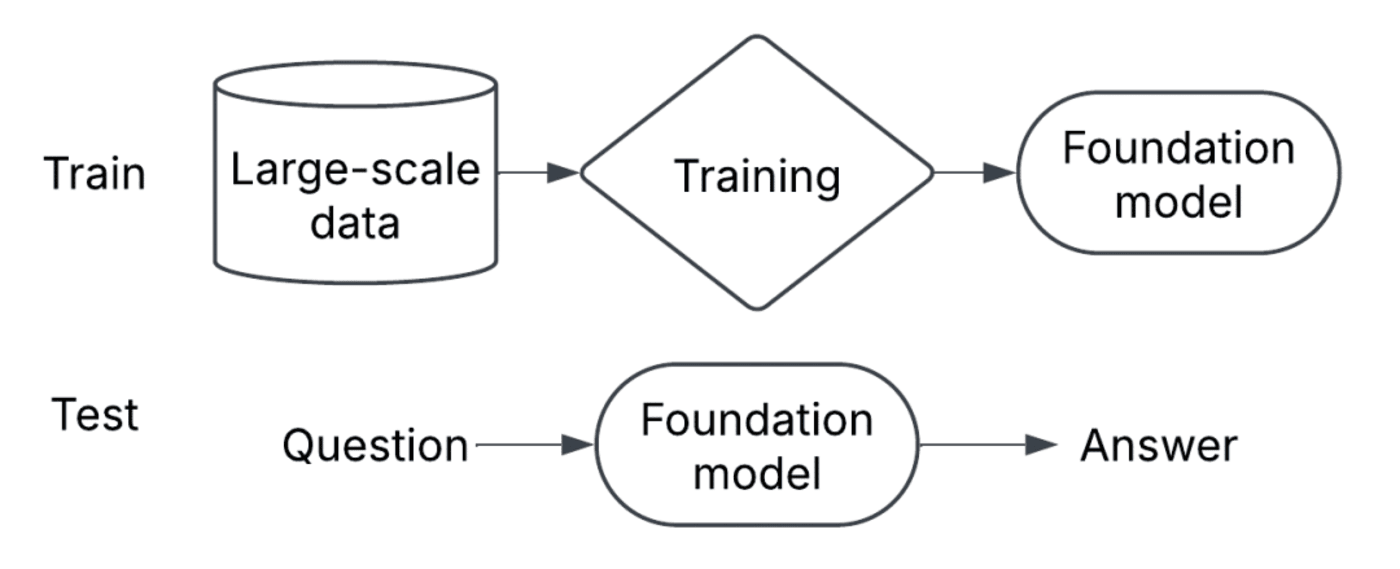
大模型(Foundation Models)是通过自监督或半监督学习,在大规模、多样化的未标注数据训练的深度神经网络模型。这些模型具备广泛的通用性,能够适应自然语言处理、计算机视觉、语音识别等多种下游任务。大模型的提出标志着人工智能系统构建方式的重大革新。
有趣的是,合成生物学的研究方法和大模型的思路不谋而合。在大模型得到完善后,合成生物学学者利用这个优势,解决了很多以往传统研究方法无法高效解决的难题,比如对于蛋白质结构的研究。然而,在神经科学领域,类似的进展尚未实现。那么,合成生物学中的经验是否可以被应用到神经科学的领域?大模型是否能够帮助神经科学突破过往的界限,大放异彩?
或许你对大模型的机制并不熟悉。那么让我们来举一个大模型中最广为人知的例子:语言模型(Language Model, LM)。语言模型就是专门处理自然语言任务的大模型。但是这一框架可以拓展到除了自然语言之外的其他范式(paradigms)中,在语言模型的语境中,任何可被离散化为“词元(token)”的数据类型均可纳入框架之中。
比如,ESM(Evolutionary Scale Modeling)是一种研究蛋白质的语言模型,通过将氨基酸残基视为词元,然后学习序列规律来建模蛋白质的功能。此外,像 HyenaDNA这样的基因组语言模型,能够处理百万级别碱基长度的DNA,并从中提取复杂信息;还有如Evo 2这样的多模态模型,能够同时理解DNA、RNA 和蛋白质的跨模态关系。
这种可迁移性来源于蛋白质结构和语言结构的高度相似性。语言的特征有很多,但是其中最相关的几种有:任意性(Arbitrariness)、可交替性(Interchangeablility)、创新性(Productivity)、离散性(Discreteness)。具体来说,我们说的每一句话都是单个要素(比如音素,语素)根据人为制定的规则形成的有限组合。而蛋白质的形成也十分类似。
然而,这种思维模式或许并不完全适用于将基础模型用于神经科学的情境——它过于聚焦于某一个庞大、单一的模型本体(如语言模型范式),而忽略了支撑其运作的整个生态系统(如标准化的数据平台)。我们不妨来深入剖析一下这个问题。
▷大模型机制的简略示意图。 @ Patrick Mineault
01 大模型与合成生物学
蛋白质对大部分人来说都是一个相当日常的概念,比如现在常说的“高蛋白低碳水饮食”。但科学界对蛋白质的理解仍然不足,就算在21世纪的今天,蛋白质仍是合成生物学和计算生物学的核心研究对象。
蛋白质的研究挑战主要源于其序列和空间结构都包含着巨大的不确定性。蛋白质由20种氨基酸组成,而即使是一条长度为100的肽链,其理论上可能的序列组合数就达到20^100,远远大于宇宙中原子的总数。除了序列排列组合的多样性,每一条氨基酸序列,都有可能折叠成不同的三维结构。所以,科学家们不仅要关注蛋白质的氨基酸序列,还要关注它们的三维结构、生物活性以及与其他分子的相互作用。
获得诺贝尔奖的AlphaFold项目的重要之处就在于,它可以根据蛋白质的序列精准预测其三维结构。这项技术的突破,得益于过去几十年科学家通过冷冻电镜(cryo-EM)、X射线晶体学等技术对蛋白质结构的细致研究,并将这些研究成果汇集于如PDB(蛋白质数据库)等的大型数据库中,使这些数据成为训练机器学习模型的基础。
但蛋白质折叠预测仅是当前生物大模型应用的冰山一角。如今该领域已扩展至更多维度。这些模型正在让我们以前所未有的方式理解生命分子,推动合成生物学进入一个由大模型驱动的新阶段。
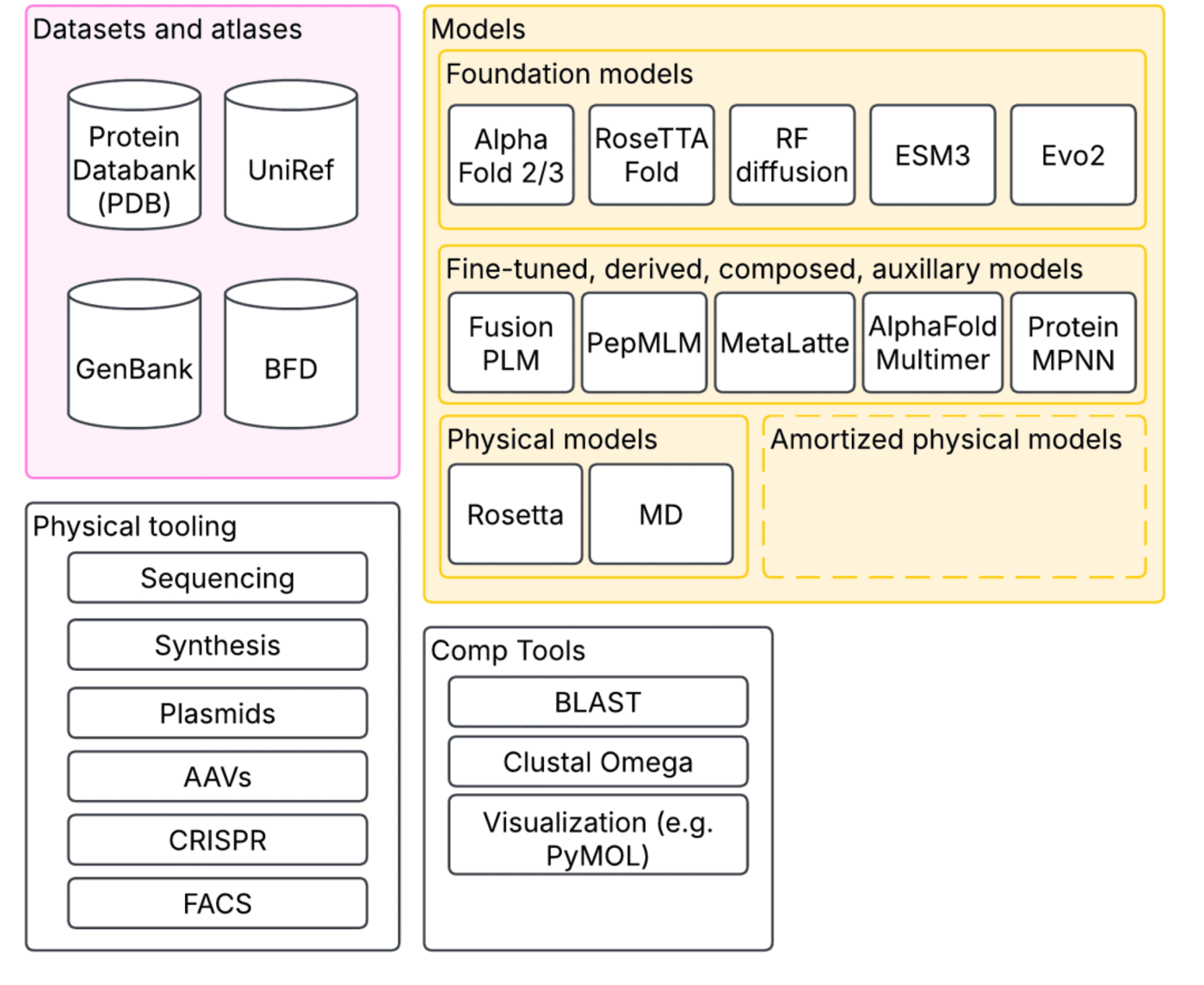
▷合成生物学中一种以生态系统为中心的大模型视角:模型只是更大生态系统中的一部分。@ Patrick Mineault
在大模型的支持下,我们不仅可以预测单个蛋白质的结构。一些模型,比如AlphaFold Multimer,能模拟多个蛋白质之间的相互作用;还有RFDiffusion,可以根据功能需求“定制”蛋白质;又比如ProteinMPNN,它则实现了“逆向折叠”,从结构反推最有可能的氨基酸序列。此外,还有许多在ESM等大模型基础上微调而来的新模型,能跨多个生物领域预测蛋白质活性。
这些技术进步依赖于长期积累的生态系统支持。数十年来积累的大量开源工具和数据库,使我们能够轻松浏览蛋白质及其序列,比对相似结构,查看三维结构……这些数据库中收录了无数蛋白质的结构、序列、生物实验数据与预测结果。
而传统的物理建模工具如Rosetta、分子动力学模拟等并没有被取代,它们仍是对机器学习方法的重要补充。更重要的是,物理模型有时还能反过来为机器学习模型提供训练数据,实现“摊销式推理”(amortized inference),即提前习得对新任务快速做出判断的能力。
在这些数十年积累的基础上,我们已突破单纯理解蛋白质的阶段,实现了对生物分子的主动改造。我们可以合成新的蛋白质序列、将其装入质粒中复制、打包进腺相关病毒(AAV)进行递送、使用CRISPR进行基因编辑、再通过测序验证编辑结果。这些技术链条的整合,大大加速了合成生物学和计算生物学的研究进程。
当然,现有的模型并非完美,但它们能通过计算筛选潜在候选方案,显著减少传统实验中耗时费力的物理优化步骤。生物学中早就习惯使用“替代终点”(surrogate endpoints),比如用小鼠模型模拟人类疾病;只是这些替代物往往存在偏差,难以直接转化为人体应用。理想的替代指标需同时满足快速和准确的要求。
举个最新的例子:Science期刊最近报道的EVOLVEPro系统展示了蛋白质设计的创新路径。蛋白质设计面临一个组合爆炸问题——对于一条有100个残基的肽链,其可能序列组合数量高达20的100次方,远超宇宙中原子的总数,因此穷举搜索完全不可行。
▷Kaiyi Jiang et al. ,Rapid in silico directed evolution by a protein language model with EVOLVEpro.Science387,eadr6006(2025).DOI:10.1126/science.adr6006。图源:Science
EVOLVEPro提供了一种高效的解决方案。它在12个深度突变扫描数据集的基准测试中(涵盖抗原结合、核酸结合、酶催化等任务)超越零样本(zero-shot)方法,并成功改造六类蛋白质,包括将单克隆抗体结合亲和力提升40倍、CRISPR核酸酶活性提高五倍。
听起来非常惊人,对吧?其实它的原理并不复杂:首先在蛋白质大模型上附加“预测头”,用已有突变数据训练其预测目标指标;随后预测出一批最有潜力的蛋白质序列,并进行合成、实验验证;再将实验结果反馈到模型中进行迭代更新,进入下一轮预测与实验。如此反复几轮,便能逐步迭代出性能更优的蛋白质。
▷EvolvePro系统示意图,摘自论文图1。PLM:蛋白质语言模型,此处为ESM2。Domain Expert Top Layer为随机森林模型
该模型基于一种蛋白质语言领域的大模型ESM2。ESM2是一种类似BERT的“掩码语言模型”(masked language model),它将蛋白质序列中的每一个残基(氨基酸)编码为一个高维向量,并通过在UniProt上的训练获得了广泛的通用性。
一旦获得蛋白质序列的向量表示(embedding),就可以被用于多种任务,比如结构预测(如ESMFold),或者可以通过对所有残基表示进行平均,得到一个固定长度的“摘要向量”。熟悉大语言模型的读者们可能会敏锐地意识到,这些摘要向量实际上就等同于 LLM中的词语向量(Word Vector)。一旦实现了对这些看似无序且无限的粒子的向量化,就可以较为轻易地在此基础上进行进一步的学习和数据处理,比如进行检索增强生成(RAG)、推荐系统、聚类分析和性质预测等任务。
在这个平均表示的基础上,研究人员还添加了一个非常简单的“预测起始站(prediction head)”——一个随机森林模型。为了获得初始数据,研究者合成了一批随机突变的蛋白质,然后进入了一个主动设计过程。他们使用模型预测下一个最值得尝试的突变,选择过程很直接:他们模拟所有可能的单残基突变,然后挑选出模型预测得分最高的前N个候选序列。
听起来很简单?的确如此。一旦拥有一个功能强大的大模型,再配合一个基础的回归器模型和一个可以快速完成“实验反馈闭环”的设计流程,优化蛋白质结构就不再遥不可及。
所有这一切的实现,要依赖一个工具与资源高度集成的生态系统:我们不仅能够随时读取和写入序列,拥有开放的数据共享平台来支持再训练和微调模型框架,还有高效的实验室自动化流程,让蛋白质活性检测变得前所未有地高效。
这标志着蛋白质优化速度发生了质的飞跃,而驱动力来自整个生态系统的协同:除了大模型,还包括数据集、数据库、结构图谱、计算工具,甚至是经典的物理建模方法。
▷用于优化GECIs的闭环系统。Wait et al.,2023年
这种技术革新对神经科学领域的影响也不容小觑。神经科学研究中对蛋白质的需求极高,从测量神经活动到精确干预神经功能,几乎每一个关键工具都依赖于蛋白质设计。比如:遗传编码的钙/电压指示器(GECIs、GEVIs),光遗传学(optogenetics),化学遗传学工具(如DREADDs等),又或者是用于神经环路映射的条形码技术(如MAP-seq、BRIC-seq、Connectome-seq)等。
更不用说当下最热门的脑部药物也是改造后的肽类分子:比如GLP-1受体激动剂,它们不仅能调节血糖、控制体重,还展现出治疗上瘾、阿尔茨海默病(AD)和帕金森病(PD)等神经退行性疾病方面的巨大潜力。
02 从合成生物学到神经科学?
那么,合成生物学中的经验能否迁移到神经科学领域呢?现阶段人类对于大脑的认知是否足以支持这种举一反三?
(1)数据是否足够?结构是否合理?
合成生物学的技术进步很大程度上是有像PDB(蛋白质数据库)和UniProt这样的高质量数据库为大模型的训练提供了坚实基础。这些数据库不仅覆盖全面,而且具有高度的注释和标准化格式,使得大规模学习变得可行。
尽管对于神经科学而言,也有一些主流的数据平台,例如DANDI、OpenNeuro,以及Allen Institute、IBL、HCP 等机构提供的高质量大脑活动数据。这些数据涵盖了大量的神经记录形式(spikes、LFP、sEEG、fMRI、EEG),总量可能超过了 10000 小时,凝聚了无数研究者的努力。
但问题在于,这些数据能否构成一个真正的“脑图谱”?目前我们仍很难同时获得具有高空间覆盖(全脑记录)、高空间分辨率、以及高任务多样性的完整数据集。这是因为现有的神经记录形式都各有侧重。比如临床常用的 fMRI 和 EEG,只能保证空间分辨率(fMRI)和时间分辨率(EEG)其中之一。而有些技术虽然可以同时保证高空间覆盖,空间分辨率和时间分辨率(如MEG),但是成本太高,并且可移动性差。就算我们拥有了高维度记录,它们也只能覆盖神经活动空间中的极小一部分。类比来说,这就像只从酵母菌的一条染色体中采样序列来训练DNA语言模型,其通用性显然受限。
细胞类型图谱、FlyWire(果蝇全脑连接组)等更“生物本位”的神经科学图谱,或许在覆盖性上更为完善,但它们仍缺少关键的跨尺度数据桥梁。例如,FlyWire 提供了果蝇脑的完整连接组,但还需要每个神经元的转录组背景、受体分布以及电生理特征来可靠地模拟神经活动。
这类桥梁就像神经科学领域的 “PDB+UniProt”,是将结构数据如连接组转化为功能机制(activity dynamics)所必需的。神经科学要构建大模型驱动的生态系统,未来十年最关键的任务之一,就是为与人类亲缘关系近的物种,甚至是人类本身,构建类似的全层次整合图谱,这包括遗传背景信息,细胞图谱与转录组数据,分子注释的中尺度与微尺度连接组,神经活动图谱,不同模态之间的“校准数据集”(即跨模态桥梁)等。
(2)我们能否主动干预大脑系统?
相比可以随意合成的蛋白质,当前神经系统的可操作性远低于蛋白质合成技术。当前带宽与维度最高的干预方式主要集中在感官外围,例如视网膜植入,或屏幕、耳机提供的视听输入。
Science Corp的生物混合器件(biohybrid devices)则采用新策略:将神经元培养在微电极与微型 LED 上,主动与设备形成交互界面,并逐步生长入脑组织。这些技术将为更精细、维度更高的调控提供可能。
除了技术上的可操作性,对于神经系统进行人为改造的伦理问题也有待考量。毕竟,我们并不希望身不由己地成为《爱,死亡与机器人》中的齐马,变成一个彻头彻尾的机械人。
▷https://science.xyz/technologies/biohybrid/
(3)能否闭环?
合成生物学中最重要的能力之一是“闭环优化”,也就是从模型预测到实验验证,再到再训练模型,最后再次优化的过程。神经科学是否实现类似流程?目前已有一些案例展现了闭环可能性的雏形,比如视觉神经元刺激中出现的“Inception loop”实验,能够寻找激活特定视觉神经元的最强刺激;以及全息光遗传学干预实验,也展示了对神经活动的主动微调。
这些实验目前仍属先驱者,但它们揭示了大模型在闭环控制中的潜力。特别是其可微特性,意味着我们可以用梯度下降等方法直接搜索最优输入刺激;但该路径依赖配套硬件的发展,仍需同步突破高精度神经记录与干预技术瓶颈。
▷V4区神经元响应的最大化刺激由Inception环路确定。 Willeke et al. (2023)
03 未来该做什么?
读到这里,亲爱的读者,您或许也已察觉:我们在神经科学中抱有的雄心——治愈所有神经类疾病、彻底理解智能与意识等——与我们目前所拥有的工具和数据,仍存在巨大落差。
大模型为我们提供了一个突破口:它们能够利用已有和未来的大规模神经数据,学习有效的神经表征,做出预测,并在闭环系统中进行优化,借助深度学习模型的可微特性实现自动调整与反馈。
但这一切不能在真空中发生。技术突破依赖数据与工具的协同发展,我们所收集的数据,以及用于“读写”神经活动、细胞类型、突触和连接的工具生态,必须彼此协同、互为促进,形成一个良性循环。要实现这一点,几乎可以肯定的是,我们需要开展大规模、非假设驱动的神经科学研究,聚焦于工具与数据本身的建设。这种研究可能会以“聚焦型研究组织”(Focused Research Organizations, FROs)或跨机构协同项目的形式展开。
非假设驱动的科学有时会被视为“无头苍蝇”,但我认为它最有力的辩护来自“认识论上的谦逊”:面对数十亿神经元、万亿级突触连接、成千上万的细胞类型、数百个脑区和受体——也许我们首先该做的,就是完成全面系统的数据采集,然后再考虑如何对其进行精准干预,从而建立真正具备因果解释力的神经模型。
未来十年,我们将拥有更多类型的神经图谱和数据库,它们将进一步支撑专用大模型与生物物理仿真模型的发展。这些知识架构将通过下一代神经技术实现互证性校准,形成动态互联的认知网络。
原文链接:
https://www.neuroai.science/p/what-are-foundation-models-for-lessons